還記得[Day 09] 提及的品質控制(Quality control)嗎?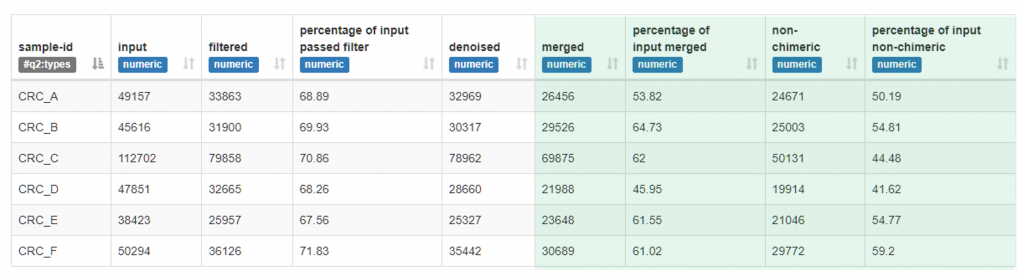
在範例的檔案中,可以發現六個樣本經過篩選條數如下 :
(其實就是上圖中最右邊的條數)
| 樣本名 | 篩選後條數 |
|---|---|
| CRC_A | 24671 |
| CRC_B | 25003 |
| CRC_C | 50131 |
| CRC_D | 19914 |
| CRC_E | 21046 |
| CRC_F | 29772 |
例如我們可以發現 CRC_D 19914 條數略少於其他組。
而假設今天樣本 N=100 分成四組,總有一些定序後條數明顯少於其他樣本,
因為後續將進行物種多樣性的分析統計等,深怕影響最終結果,
希望能將一些樣本去除,畢竟條數少可能造成偏差(bias)。
如果你是生科相關科系學生,在做蛋白質定量法繪製標準曲線(standard curve)時,
若做了蠻多個點,可能也會刪去幾個讓R^2結果更漂亮 (笑 :
Reference : ZGENEBIO BIOTECH INC.
而取樣深度 (Sampling depth) 也有異曲同工之妙,
刪去幾個各種原因造成的低序列數樣本,讓整體結果更有說服力。
這題並無標準答案,據 QIIME2 開發團隊表示 :
分析者必須在 :
留下最多的序列條數 (The most sequences)及
留下最多的樣本 (The most samples) 做平衡,
如果樣本數少、當作練習、樣本很難採集,
且條數差距不大(可能都有幾萬條),可以考慮都留下,
若樣本數多,動輒數百,則 QIIME2 提供取樣深度網頁拖拉服務,
提供切一刀的數字參考 :
先將[Day 09]得到的 table-dada2-240.qza轉換為qzv
qiime feature-table summarize \
--i-table table-dada2-240.qza \
--o-visualization table-dada2-240.qzv \
--m-sample-metadata-file sample-metadata.tsv
完成後會顯示 :
'
Saved Visualization to: table-dada2-240.qzv
'
拖曳到 QIIME2 VIEW,右側有個滑桿~
可以依據組別進行觀察 (sample-metadata.tsv中含有 Index 一人一組與 Sex),
深度取的越深,留下來的樣本就越少(紅色代表該樣本會被篩掉),
Index 個別樣本觀察。

Sex 組別觀察,可發現深度取越深,各組所剩的樣本數會開始不平均,
以範例來說,會發現 Male 組樣本數會下降較多,
所以該取多少是分析人要思考的問題。
主因是樣本數很少(N=6)很可憐了,捨不得放棄他們,
再者其實條數都算多,之後的稀疏分析(Alpha Rarefaction)會介紹,
實務上,其實有上萬條都很足夠,
但若遇到同採集條件的樣本卻只有一兩千條,就要思考要不要去除。
如果你心中叛逆的性格被挑起了,
範例檔案可以設 19915,(即只高最低的樣本1,使得N=5),不影響後續教學。
備註 : 每次QC結果剩餘的條數因演算法關係,可能略有不同,
就算同為範例檔案,也可能有個位數條數差異,
所以在這裡深度以自己跑分析的結果為主。
怎麼辦,我有選擇困難,都有上萬條我不知道誰要留阿QQ,覺得都好重要,
都留下吧! 不然跟教授解釋200個樣本下去分析怎麼剩下150個也是蠻麻煩的
取樣深度取最低的樣本序列條數 = 全部保留
(翻找好多 QIIME2 社群問答歸納的結論XD)
本篇使用到的輸入/輸出檔案 :Input : table-dada2-240.qza、sample-metadata.tsvOutput: table-dada2-240.qzv
拿起一張便條紙記下 Sampling depth,
以及最大的序列條數 (範例是:50131) 之後會使用到~
本篇文章同步刊載於科學毛怪部落格 PetSci Blog。
